Овцебык, или мускусный бык (Ovibos moschatus), – растительноядное животное, единственный современный представитель рода овцебыков из семейства Полорогих. Дальние предки современных овцебыков вместе с бизонами, мамонтами и шерстистыми носорогами обитали в высокогорьях Центральной Азии 3–4 млн лет назад [1; 2]. В настоящее время считается, что систематическое положение овцебыка окончательно не определено.
Цель исследования: анализ современного состояния систематической принадлежности Ovibos moschatus в филогенетическом древе на основе литературных данных из работ в области молекулярной генетики.
В систематику овцебык был введен как бык мускусный (Bos moschatus) и отнесен к роду быков (Bos) подсемейства бычьих (Bovinae) семейства Полорогих (Bovidae) Циммерманом в 1780 г. В связи с тем что наряду с внешним сходством с быками у Bos moschatus имелись анатомические сходства с баранами, в начале XIX века Де Блэнвил отделил овцебыка от рода Bos и создал для него монотипный род Оvibos. Затем Аллен (1913) вновь включил этот род в подсемейство бычьих, так как отмечал близкое родство овцебыка с бизонами. В настоящее время в систематике зоологи придерживаются классификации Г. Симпсона (1945), который овцебыка включил в подсемейство Caprinae (козлов и баранов) и выделил в его пределах трибу Ovibovini (рис. 1) [3].
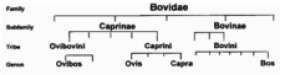
Рис. 1. Таксономическая связь между овцебыками и другими представителями семейства Bovidae, по Якушкину, 1998 [3]

Рис. 2. Филогенетическое древо гена цитохрома b в митохондриальной ДНК, показывающее взаимоотношения пяти видов Caprinae. Длина ветвей приведена с верхней стороны веток. Значения начальной загрузки (основанные на 1000 повторностях) приведены с нижней стороны веток. Корова была включена как внешняя группа. (a) – значения представляют три различных анализа с использованием информативных сайтов / с исключенными переходами третьей позиции только с сайтами трансверсии. Длина этих ветвей составляла 259, 104 и 48 соответственно. CI были 0,64, 0,71 и 0,77, а g1 – 20,77, 20,96 и 20,87 соответственно; (б) – значения расстояния древ для максимального правдоподобия. Длины ветвей даны как 10-кратное процентное значение (по Гровс и др., 1997) [7]
Эркхарт (Urquhart D.R.) в своей книге «Muskox: life history and current status of muskoxen in the N.W.T» [4] в 1982 г. обратил внимание на отличительные особенности овцебыка по количеству хромосом, обнаруженных при анализе кариотипов представителей семейства полорогих. Он отличается по диплоидному числу хромосом и от крупного рогатого скота (2n = 60), и от овец (2n = 54), а при этом сходен с буйволом (2n = 48), хотя относится к подсемейству Caprinae (козлов и баранов), так же как и его исторические предшественники [4].
Предковые формы овцебыков – группа родов Ovibovini жили еще в миоплиоценовое время, впоследствии они вымерли, но дали новые виды [5]. Прямым предком овцебыка считается Bootherium bombifrons (шлемоносный мускусный бык) из раннего плиоцена. В августе 2018 г. Пер Бовер (Pere Bover) и др. в журнале «Molecular Phylogenetics and Evolution» опубликовали статью «Molecular resolution to a morphological controversy: The case of North American fossil muskoxen Bootherium and Symbos». Ископаемые виды Bootherium (шлемоносный мускусный бык) и Symbos систематики считали разными видами, но были предположения, что это один вид с половым диморфизмом. С помощью генетических анализов семи ископаемых материалов Пер Бовер (Pere Bover) с коллегами смогли подтвердить, что это действительно один вид овцебыков. Они также доказали с помощью филогенетического анализа, что род Ovibos и род Bootherium являются родственными, и их пути разошлись около 3,2 млн лет назад [6].
В 1997 г. Памела Гровс и Геральд Шилдс опубликовали статью «Cytochrome B Sequences Suggest Convergent Evolution of the Asian Takin and Arctic Muskox», в которой они попытались установить филогенетическое положение и родство такинов и овцебыков с помощью анализа последовательностей цитохрома b в мтДНК. Результаты этих исследований приведены на рис. 2. Из рисунка видно, что пути такина и овцебыка разошлись довольно-таки давно и генетически эти животные уже сильно отличаются, но, несмотря на это они, имеют общего предка [7].
В 2009 г. в статье «Evolution of the Mitochondrial Genome in Mammals Living at High Altitude: New Insights from a Study of the Tribe Caprini (Bovidae, Antilopinae)» описаны результаты, полученные Краурд и др. при анализе филогенетических отношений представителей подсемейства Caprinae. Они провели исследование митохондриального генома, в частности участка D-петли. Результаты этих исследований даны на рис. 3 [8].
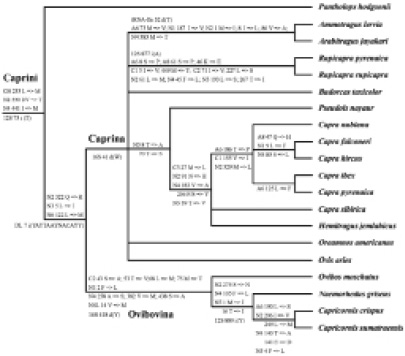
Рис. 3. Диагностические изменения аминокислот и инсерции. Картирование молекулярных сигнатур (негомопластических превращений), соответствующих либо аминокислотным изменениям, обнаруженным в 13 кодирующих белок генах (A6, A8; CB; C1, C2, C3; N1, N2, N3 N4, N4L, N5, N6), либо инсерциям (i: вставка; d: делеция), обнаруженным в контрольной области, генах рРНК (12S и 16S) и тРНК. Позиции инсерций и делеций обозначены со ссылкой на последовательность Ovis Aries (NC_001941) (по Кроурду и др., 2009) [8]
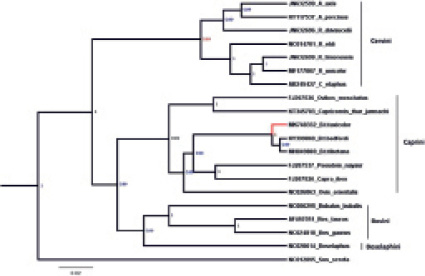
Рис. 4. Филогенетические отношения между такином и родственными видами, выведенными из 13 PCG и двух рРНК с использованием байесовского вывода (BI). Байесовские апостериорные значения вероятности показаны в каждом узле. Sus scrofa (NC012095) использовали в качестве внешней группы (по Кумару и др., 2019) [9]
В сентябре 2019 г. Аджит Кумар и др. опубликовали статью «Sequencing and characterization of the complete mitochondrial genome of Mishmi takin (Budorcas taxicolor taxicolor) and comparison with the other Caprinae species» в журнале «International Journal of Biological Macromolecules». В этой статье авторы сравнили генетический материал по мтДНК такинов, овцебыков, а также других представителей подсемейства Caprinae. Результаты исследований приведены на рис. 4 [9].
В октябре 2005 г. Росс Де Макфи (Ross DE MacPhee) и др. опубликовали статью «Late Quaternary loss of genetic diversity in muskox (Ovibos)». Они провели генетический анализ ископаемого материала останков древних овцебыков, живших с позднего плейстоцена до позднего голоцена, найденных на территории местностей Северо-Восточной Азии и Арктического архипелага северной части Северной Америки, и сравнили с современным овцебыком. В своей статье они пришли к выводу, что Ovibos moschatus был генетически более разнообразным в позднем плейстоцене, чем сегодня [10; 11]. Современные овцебыки по сравнению с другими видами полорогих имеют самое низкое генетическое разнообразие по ядерным и митохондриальным генам и характеризуются низкой полиморфностью микросателлитных локусов. В результате изолированности популяций, а также их маленьких эффективных размеров в настоящее время отмечается высокий уровень гомозиготности генома овцебыка, что может приводить к потерям генетической изменчивости. По теории популяционной генетики снижение генетической изменчивости в результате генетико-автоматических процессов в малых популяциях ограничивает адаптационные способности популяции и увеличивает зависимость их размеров от случайных колебаний окружающей среды и вероятность вымирания в изменчивых условиях среды [12; 13].
В 2015 г. в журнале BMC Evolutionary Biology опубликована статья «Looking for adaptive footprints in the HSP90AA1 ovine gene», в которой авторы проверили наличие и сравнили аллельные частоты полиморфизмов гена HSP90AA1 среди одомашненных и диких овец и представителей подсемейства Caprinae. Они выяснили, что полиморфизмы гена HSP90AA1, обнаруженные у видов Ovis aries, также присутствуют у диких видов из подсемейства Caprinae (в том числе и овцебыка), что указывает на более древнее происхождение этих мутаций и ее важность в адаптации видов к прошлым климатическим условиям естественной среды их существования [14].
В 2013 г. в журнале «Известия ТСХА» была опубликован статья В.И. Глазго и др. «Биомаркеры геномной нестабильности у животных сельскохозяйственных видов». В своей работе они выполнили сравнительный анализ частот встречаемости эритроцитов с микроядрами в клетках периферической крови ряда сельскохозяйственных видов млекопитающих и диких овцебыков. Частота встречаемости эритроцитов с микроядрами оказалась выше (Р < 0,05) у доместицированных видов по сравнению с овцебыком. Таким образом, наблюдается определенная тенденция к относительно повышенной геномной нестабильности у доместицированных животных по сравнению с диким видом [15].
В 2007 г. при работе с современными и древними ДНК овцебыков ученые «Института сравнительной геномики Саклера» обнаружили, что у овцебыков, как современных, так и древних, имеются митохондриальные вставки в ядре (псевдогены). В статье «Detection of mitochondrial insertions in the nucleus (NuMts) of Pleistocene and modern muskoxen», опубликованной в том же году, Гринвуд (Greenwood A.D.) и др. отмечают, что это открытие ставит под сомнение точность получаемых данных при ПЦР и секвенировании ДНК. Но и в то же время они разработали несколько методик, которые позволяют получить более или менее достоверные данные, хотя на результаты сильно влияет качество исследуемых образцов, из которых выделяется ДНК [16].
В 2015 г. Бардуков Н.В. выступил на VIII Московском международном конгрессе РХТУ им. Д.И. Менделеева с докладом «Древние и современные геномы Ovibos moschatus» [17]. По результатам сравнительного анализа генетической структуры современных реинтродуцированных популяций овцебыка с ископаемыми представителями вида по полилокусным ДНК-маркерам он выявил наличие «консервативной» (встречается у всех, включая ископаемых представителей) и «вариабельной» частей генома [18]. А также обнаружил отсутствие суммарно по всем маркерам ISSR и IRAP 30 локусов у современных овцебыков и наличие у них только 14 «новых» локусов, что может быть также подтверждением гипотезы о наличии большего генетического разнообразия у предков овцебыков. Кроме того, по результатам генотипирования по классу хелитронов-ДНК-транспозонов, способных реплицироваться по типу «катающегося кольца», были получены высоко консервативные спектры. Найденные консервативные локусы могут быть использованы в эволюционной геномике при сравнительном анализе древних и современных популяций животных. У современных животных обнаружено 14 локусов, имеющих высокий полиморфизм в инбредных популяциях, что может быть следствием активации транспозиций МГЭ (мобильных генетических элементов) [18].
В 2011 г. Бардуков и др. опубликовали статью в журнале «Известия Тимирязевской сельскохозяйственной академии» [19], в которой приводят результаты сравнительных анализов генетической структуры животных трех популяций овцебыков, обитающих в Восточной Гренландии, на полуострове Таймыр и острове Врангеля, по двум методам – ISSR-PCR и IRAP-PCR. Полученные по двум методам данные были не однозначные, но в целом они приводят к выводу, что гренландская популяция овцебыков имеет наименьшее сходство генетической структуры с таймырской и врангелевской популяциями. Хотя по IRAP-маркерам к гренландской популяции более ближе таймырская, нежели врангелевская популяция. Основные отличия были по доле полиморфных локусов и расчитанным индексам полиморфного информационного содержания каждого локуса (PIC). Результаты генетической характеристики популяций, полученные с помощью ISSR-маркеров, не были идентичными результатам, полученным методом IRAP-PCR, но полученные дендрограммы генетических взаимоотношений по результатам обоих методов были идентичны, качественно отличались от выявленных с помощью ISSR-маркеров, за исключением кластеризации популяций при построении дендрограмм генетических взаимоотношений (рис. 5, 6).
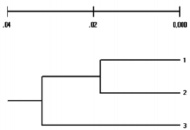
Рис. 5. Дендрограмма генетических взаимоотношений между популяциями, построенная по ISSR-маркерам: 1 – популяция полуострова Таймыр; 2 – популяция острова Врангеля; 3 – нативная популяция Восточной Гренландии (по Н.В. Бардукову и др., 2011) [19]
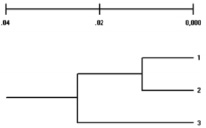
Рис. 6. Дендрограмма генетических взаимоотношений между популяциями, построенная по IRAP-маркерам: 1 – популяция полуострова Таймыр; 2 – популяция острова Врангеля; 3 – нативная популяция Восточной Гренландии (по Н.В. Бардукову и др., 2011) [19]
В этих исследованиях полученные генетические дистанции, рассчитанные по ампликонам, полученным с праймерами PawS 5 и LTR-SIRE-1, показывают расхождение с теорией происхождения популяций овцебыков (табл. 1, 2) [19].
Таблица 1
Доля полиморфных локусов в спектрах амплификации ( %) (по Н.В. Бардукову и др., 2011) [19]
|
Праймер |
Таймыр |
Врангель |
Гренландия |
По всем |
|
(AG)9C (GA)9C (GAG)6C LTR SIRE-1 PawS 5 |
8,33 42,86 16,67 41,67 14,29 |
0,00 42,86 8,33 41,67 14,29 |
16,67 35,71 16,67 8,33 0 |
25,00 50,00 25,00 50 14,29 |
Таблица 2
Полиморфное информационное содержание спектров продуктов амплификации (по Н.В. Бардукову и др., 2011) [19]
|
Праймер |
Таймыр |
Врангель |
Гренландия |
По всем |
|
(AG)9C (GA)9C (GAG)6C Праймер LTR-SIRE-1 Праймер PawS 5 По ISSR-праймерам По ретротранспозонам |
0,04 0,13 0,06 0,18 0,05 0,08 0,13 |
0,00 0,14 0,03 0,18 0,07 0,06 0,14 |
0,07 0,16 0,07 0,04 0,00 0,10 0,02 |
0,07 0,18 0,08 0,18 0,06 0,11 0,13 |
В 2012 г. этими же авторами опубликована еще одна статья «Polymorphism of ISSR and IRAP markers in genomes of Musk-oxen (Ovibos moschatus) and horse (Equus caballus) of Altaic breed» [20]. В этой статье Глазко и др. сравнили по ISSR-PCR и IRAP-PCR маркерам полилокусы трех популяций овцебыков из Восточной Гренландии, с полуострова Таймыр и острова Врангеля и алтайской породы лошадей. Они обнаружили, что уровень полиморфизма большинства маркеров у лошади был значительно выше, чем у овцебыков. Были выявлены различия в характеристиках полиморфизма маркеров ISSR-PCR и IRAP-PCR между видами. А также выявлено, что видоспецифичные сегменты ДНК, окруженные инвертированными повторами микросателлитов, могут образоваться в результате рекомбинации между эволюционно более «древними» и «молодыми» мобильными генетическими элементами.
В октябре 2019 г. Graphodatsky A.S., Проскурякова А.А. и др. [21] на сайте «Национальный центр биотехнологической информации (National Center for Biotechnology Information)» в разделе Национальной медицинской библиотеки США опубликовали статью «Сравнительное хромосомное картирование овцебыка и Х-хромосомы у некоторых видов Bovidae». В этой статье представлена детальная сравнительная карта хромосом овцебыка (рис. 7).
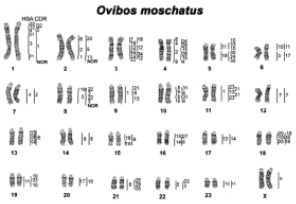
Рис. 7. Сравнительная карта хромосомной окраски овцебыка с гомологиями к человеческому (HSA) и дромедарию (CDR). Нуклеолярные организующие области (NOR) показывают локализацию нуклеолярной организующей области (по Graphodatsky A.S. и др., 2019) [21]

Рис. 8. Гомология хромосом видов Pecoran (PAK) с хромосомами родового кариотипа и с человеческими гомологиями слева и дромадером справа. Представленные виды включают: AAM (Antilocapra americana), MMO (Moschus moschiferus) и представителей различных популяций Bovidae: PNG (Pseudoryx nghetinhensis), BTA (Bos taurus) (Bovini), OAR (Ovis aries), OMO (Ovibos moschatus) (Caprini)), DHU (Damaliscus hunteri) (Alcelaphini). Центромеры отмечены звездочкой. Новые данные, полученные в исследовании, отмечены черным кружком в углах клеток (по Graphodatsky A.S. и др., 2019) [21]
По этой карте кариотип овцебыка включает 6 субметацентрических и 17 акроцентрических аутосом и одну пару половых хромосом (2n = 48). Основное количество аутосомных плеч у овцебыка 58, что в целом характерно для кариотипов семейства Bovidae. По результатам этой работы овцебык является представителем подсемейства Antilopinae, трибы Caprini. Как для карты сравнительного сцепления, так и для карты FISH показано одно существенное различие между кариотипами овец (Antilopinae, Caprini) и крупного рогатого скота (Bovinae). Это различие произошло из-за транслокации с участием сегментов, гомологичных BTA 9 и BTA 14. Однако эта связь не наблюдается у овцебыков. Предположительно, это определяется базальным положением Ovibovina, что позволяет предположить, что транслокация BTA 9/14 произошла только в субтрибе Caprina (рис. 8) [21].
Заключение
Маскоксены (Ovibos moschatus) являются неотъемлемым компонентом биоразнообразия Арктики. Учитывая низкое генетическое разнообразие, их способность реагировать на будущие и быстрые изменения в Арктике неизвестна, хотя палеонтологическая история демонстрирует адаптивность в определенных пределах [22].
В настоящее время интерес к филогенетическим исследованиям растет. В результате современных исследований происходит пересмотр некоторых представлений о происхождении и родственных связях разных видов животных и растений. Традиционными методами филогенетических исследований являются сравнительно-морфологический, палеонтологический, онтогенетический, а в настоящее время с развитием молекулярной биологии эффективность и точность исследований резко возросла. В частности, очень перспективное открытие сделали Бакли и др. в 2010 г. Они идентифицировали пептидную последовательность коллагенового типа I (COL1A2), которая может помочь в изучении филогенетических отношений внутри подсемейства Caprinae [23]. Это открытие уже использовалось и доказало свою эффективность для изучения филогении семейства Bovidae, но только для одомашненных представителей [24]. Также многими авторами были описаны и уже используются новые методики для работы с деградированной древней ДНК, в частности можно отметить работы Гилберта и др. [25; 26].
Таким образом, активно ведутся работы по изучению генома овцебыка, по отдельности были изучены многие генетические аспекты, например такие, как хромосомный анализ современных и вымерших овцебыков внутри вида, степень их родства. Но, несмотря на это, до сих пор не разрешен вопрос: от какого подсемейства произошел овцебык, от подсемейства бычьих или от подсемейства козьих? В связи с последними достижениями науки, развитием технологий, созданием новых методик изучения ДНК этот спорный вопрос решается на уровне генетических исследований.


 science-review.ru
science-review.ru