Введение
Главный комплекс гистосовместимости (ГКГ) представляет собой область генома, содержащую гены, критически важные для иммунного ответа. Классические гены ГКГ кодируют специализированные гликопротеины, выполняющие функцию презентации антигенов – как эндогенных, так и экзогенных – на клеточной поверхности для распознавания Т-лимфоцитами и NK-клетками. Наиболее примечательной характеристикой генов ГКГ является их исключительный полиморфизм: в природных популяциях описаны десятки и даже сотни аллельных вариантов. Максимальное разнообразие наблюдается в пептид-связывающем домене PBS (peptide binding sites), особенно в участках непосредственного контакта с антигеном. Эволюционный анализ свидетельствует об адаптивной природе этого полиморфизма, доказательством являются преобладание несинонимичных замен в PBS, повышенные значения коэффициентов отбора и сохранение аллельного разнообразия в течение длительного эволюционного времени. Уникальное сочетание высокого полиморфизма с породоспецифичным распределением аллелей делает гены ГКГ идеальной моделью для изучения механизмов поддержания генетического разнообразия в популяциях под действием естественного отбора [1].
Гены главного комплекса гистосовместимости человека HLA подразделяются на три основных класса и локализованы на 6-й хромосоме. Гены класса I ответственны за презентацию антигенов, локализованных в цитоплазме, гены класса II осуществляют презентацию антигенов, находящихся во внеклеточной среде или внутриклеточных везикулах, тогда как гены класса III вовлечены в процессы воспалительного ответа [2].
Сходство иммунных систем человека и млекопитающих, включая развитие аналогичных патологий при их нарушениях, делает животных ценными моделями для исследований. Домашняя собака Canis lupus familiaris является одним из наиболее перспективных видов для таких исследований. Изучение генов главного комплекса гистосовместимости собаки DLA (dog leucocyte antigen) относится к началу 1960-х гг. [3]. В этих работах была установлена главная биологическая роль антигенов собаки в процессе отторжения или приживания аллогенных тканевых трансплантатов [4]. Эти работы вызвали большой интерес в плане изучения главного комплекса гистосовместимости и рассмотрения собаки как модели для разработки методов трансплантации органов и тканей человека [5].
Локус лейкоцитарного антигена DLA у собак, содержащий большое количество функциональных генов иммунной системы, расположен на 12-й хромосоме [6]. DLA собак играет важную роль в системе иммунного ответа и состоит из трех районов, обозначаемых как класс I, класс II и класс III. Схема организации DLA собак представлена на рис. 1. Гены DLA принадлежат к первым двум классам и вовлечены в регуляцию участия антигенов в иммунной системе. Было идентифицировано 4 гена, входящих в класс I локуса DLA собаки. Это DLA-12, DLA-64, DLA-79, DLA-88 [7]. Для каждого из перечисленных генов обнаружено разное число аллелей, причем 3 из них, DLA-12, DLA-64, DLA-79, имеют 17, 9 и 8 аллелей, соответственно, а для гена DLA-88 описано 139 аллельных вариантов [8–10].
Структура локуса DLA класса II включает 4 гена: DRA1, DRB1, DQA1 и DQB1. ГенDRA1 является мономорфным, в то время как гены DRB1, DQA1 и DQB1 полиморфны и включают 181 DRB1, 30 DQA1 и 86 DQB1 аллельных варианта [12]. Полиморфизм локуса DLA ограничен внутри одной породы, но существуют значительные различия в аллельных вариантах и их частотах встречаемости у представителей разных пород собак [13]. Локус DLA класса III у домашних собак содержит гены ортологи системы комплемента, цитокинам и белкам теплового шока, которые полностью соответствуют по структуре и функции генам HLA класса III у человека.
Актуальность изучения ограниченного разнообразия гаплотипов генов локуса DLA класса II у чистопородных собак возрастает в связи с расширением практики породного разведения в географически изолированных популяциях, поскольку данный фактор может иметь существенное клинико-эпидемиологическое значение.
Целью исследования была оценка полиморфизма генов класса II локуса DLA среди собак пород российского разведения.
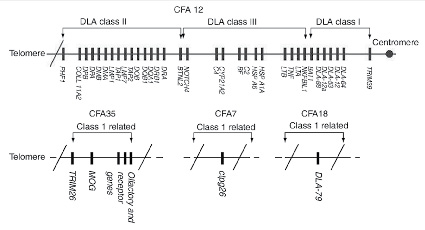
Рис. 1. Хромосомная локализация и взаимное расположение генов главного комплекса гистосовместимости DLA классов I, II, III [11]
Материалы и методы исследования
Исследование выполнено на образцах буккального эпителия, полученного от собак четырех пород российской селекции. Отбор пород для анализа проводили исходя из двух ключевых критериев: необходимости первичной молекулярно-генетической характеристики и наличия литературных данных о взаимосвязи полиморфизма генов главного комплекса гистосовместимости с иммунопатологиями у различных пород собак. Так были отобраны представители четырех пород, среди которых самоедская лайка, сибирский хаски, русский черный терьер и боксер. Для проведения исследования использовали по 12 образцов ДНК клинически здоровых особей каждой породы, предоставленных ветеринарной клиникой «Зооген». При формировании выборки соблюдали гендерный баланс, включив равное количество особей каждого пола.
Методы, примененные в исследовании: выделение геномной ДНК, амплификация и анализ генов локуса DLA, секвенирование и статистический анализ ампликонов, построение филогенетических деревьев по методу Байеса.
Выделение геномной ДНК
Экстракцию ДНК из образцов буккального эпителия проводили методом ферментативного лизиса с температурной денатурацией [14]. Протокол включал: инкубацию проб при 65 °C для лизиса клеток; сорбцию нуклеиновых кислот на силикатной матрице; серию отмывок для удаления белковых примесей; элюцию ДНК в ТЕ-буфере (10 мМ Tris-HCl, 1 мМ EDTA, pH 8.0); финальную очистку методом центрифугирования (10,000 ×g).
Амплификация и анализ генов локуса DLA
Для ПЦР-анализа были разработаны специфические праймеры, фланкирующие полиморфные регионы генов: DRB1 (322 п.н.), DQA1 (347 п.н.) и DQB1 (300 п.н.). Амплификацию проводили с универсальным Т7-праймером, рекомендованным для секвенирования высокогетерозиготных участков. Полученные ампликоны подвергали электрофоретическому разделению в 2 % агарозном геле с последующей визуализацией в УФ-свете.
Секвенирование и статистический анализ ампликонов
По завершении ПЦР-амплификации продукты реакции были направлены в Ресурсный центр СПбГУ для проведения секвенирования по методу Сенгера. Определение нуклеотидных последовательностей генов DRB1, DQA1 и DQB1 выполняли с использованием прямых и обратных праймеров и стандартного протокола терминаторного секвенирования. Для каждой из исследуемых пород было проанализировано по 12 индивидуальных ПЦР-амплификатов, что в сумме составило 48 сиквенсов на ген. Биоинформатический анализ включал визуализацию хроматограмм в программе Chromas 2.6.6, выравнивание полученных последовательностей с референсными геномами и идентификацию аллельных вариантов по базе данных IPD-MHC [15]. Для определения частот гаплотипов применяли подход, описанный Кеннеди с соавт., в котором частоту гомозиготных особей вычисляли как отношение числа гомозигот к общему количеству исследованных животных в породной группе. Частоту гетерозигот определяли аналогичным образом [10, 11, 13].
Построение филогенетических деревьев по методу Байеса
Для выявления эволюционных взаимосвязей между аллельными вариантами генов локуса DLA (DRB1, DQA1, DQB1) класса II был проведен комплексный Байесовский филогенетический анализ с использованием алгоритма, предложенного Джарвисом с соавт. [16]. Анализ включал построение отдельных филогенетических реконструкций для каждого исследуемого гена в рамках каждой анализируемой породы.
Результаты исследования и их обсуждение
По результатам секвенирования образцов ДНК, полученных от собак четырех исследованных пород: самоедской лайки, сибирского хаски, русского черного терьера и боксера – был проведен анализ аллельного разнообразия генов класса II локуса DLA (рис. 2).
При сравнительном анализе числа аллелей по отдельным генам в пределах каждой из пород следует отметить, что у самоедской лайки максимальное число аллелей, 12, отмечено для гена DQB1. Такое же число аллелей для этого гена обнаружено в породе русский черный терьер, а схожее число аллелей, 12, обнаружено для гена DRB1 в породе сибирский хаски. Эти два гена характеризуются наибольшей изменчивостью по числу аллелей, по сравнению с геном DQA1 во всех четырех породах. В частности, у самоедской лайки и русского черного терьера было выявлено 4 и 5 аллелей соответственно, а для сибирского хаски и боксера – 8 и 7 аллелей соответственно.
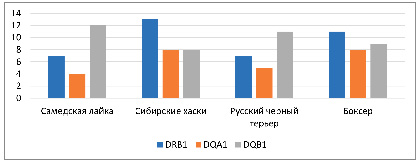
Рис. 2. Число аллелей по генам DRB1, DQA1, DQB1 у собак пород российского разведения Источник: составлено авторами
Таблица 1
Породоспецифичные гаплотипы по генам DRB1, DQA1, DQB1 локуса DLA пород самоедская лайка, сибирский хаски, русский черный терьер, боксер российской селекции (выделены характерные гаплотипы)
|
№ собаки |
DRB1 |
DQA1 |
DQB1 |
|
Самоедская лайка |
|||
|
1 |
DRB1*040:01 |
DQA1*022:01 |
DQB1*019:01 DQB1*054:02 |
|
2 |
DRB1*015:01 |
DQA1*006:01 |
DQB1*019:01 DQB1*SAM1 |
|
3 |
DRB1*015:01 |
DQA1*022:01 |
DQB1*019:01 DQB1*054:02 |
|
4 |
DRB1*015:01 |
DQA1*006:01 |
DQB1*019:01 DQB1*SAM2 |
|
5 |
DRB1*015:01 |
DQA1*006:01 |
DQB1*019:01 DQB1*SAM3 |
|
6 |
DRB1*015:01 |
DQA1*006:01 |
DQB1*019:01 DQB1*054:02 |
|
7 |
DRB1*SAM3 |
DQA1*006:01 |
DQB1*057:01 DQB1*SAM4 |
|
8 |
DRB1*001:01 DRB1*009:01 |
DQA1*022:01 DQA1*014:01:1 |
DQB1*036:01 DQB1*SAM6 |
|
9 |
DRB1*SAM1 DRB1*SAM2 |
DQA1*001:01 |
DQB1*002:01 DQB1*SAM5 |
|
10 |
DRB1*015:01 |
DQA1*006:01 DQA1*001:01 |
DQB1*019:01 DQB1*SAM7 |
|
Сибирский хаски |
|||
|
1 |
DRB1*015:01 |
DQA1*010:01 |
DQB1*019:01 |
|
2 |
DRB1*HAS1 DRB1*HAS2 |
DQA1*009:01 DQA1*010:01 |
DQB1*023:01 DQB1*020:01 |
|
3 |
DRB1*040:01 |
DQA1*010:01 |
DQB1*019:01 |
|
4 |
DRB1*040:01 DRB1*015:02 |
DQA1*022:01 DQA1*010:01 |
DQB1*019:01 DQB1*HAS1 |
|
5 |
DRB1*040:01 DRB1*001:01 |
DQA1*022:01 DQA1*HAS2 |
DQB1*019:01 DQB1*HAS2 |
|
6 |
DRB1*040:01 |
DQA1*010:01 |
DQB1*019:01 |
|
7 |
DRB1*040:01 |
DQA1*010:01 |
DQB1*019:01 |
|
8 |
DRB1*040:01 DRB1*HAS5 |
DQA1*024:01:1 DQA1*HAS3 |
DQB1*019:01 DQB1*029:01 |
|
9 |
DRB1*049:01 DRB1*HAS6 |
DQA1*024:01:1 DQA1*HAS3 |
DQB1*019:01 DQB1*HAS4 |
|
10 |
DRB1*015:01 |
DQA1*006:01 |
DQB1*023:01 |
|
11 |
DRB1*05701 DRB1*HAS3 |
DQA1*024:01:1 DQA1*HAS3 |
DQB1*019:01 DQB1*HAS3 |
|
12 |
DRB1*049:02 DRB1*HAS4 |
DQA1*010:01 DQA1*HAS1 |
DQB1*HAS4 |
|
Русский черный терьер |
|||
|
1 |
DRB1*001:01 |
DQA1*003:01 |
DQB1*004:01 |
|
2 |
DRB1*018:01 DRB1*001:01 |
DQA1*010:01 DQA1*RBT1 |
DQB1*005:04 DQB1*RBT3 |
|
3 |
DRB1*001:05 DRB1*RBT1 |
DQA1*003:01 DQA1*005:01:1 |
DQB1*048:02 DQB1*RBT1 |
|
4 |
DRB1*005:01 DRB1*006:01 |
DQA1*005:01:1 DQA1*003:01 |
DQB1*HAS1 DQB1*RBT2 |
|
5 |
DRB1*001:01 |
DQA1*003:01 |
DQB1*RBT4 |
|
6 |
DRB1*001:01 |
DQA1*003:01 |
DQB1*004:01 |
|
7 |
DRB1*001:01 |
DQA1*003:01 |
DQB1*HAS1 |
|
8 |
DRB1*001:01 |
DQA1*003:01 |
DQB1*004:01 |
|
9 |
DRB1*001:01 DRB1*020:01 |
DQA1*003:01 DQA1*004:01 |
DQB1*048:02 |
|
10 |
DRB1*001:01 |
DQA1*003:01 |
DQB1*004:01 |
|
11 |
DRB1*001:01 DRB1*006:01 |
DQA1*003:01 DQA1*005:01:1 |
DQB1*004:01 DQB1*028:01 |
|
12 |
DRB1*001:01 DRB1*006:01 |
DQA1*003:01 DQA1*004:01 |
DQB1*038:02 DQB1*RBT5 |
|
Боксер |
|||
|
1 |
DRB1*015:02 |
DQA1*006:01 |
DQB1*023:01 |
|
2 |
DRB1*015:02 |
DQA1*006:01 DQA1*BOX3 |
DQB1*023:01 |
|
3 |
DRB1*004:01 DRB1*013:01 |
DQA1*014:01:1 DQA1*BOX4 |
DQB1*015:01 DQB1*002:01 |
|
4 |
DRB1*015:02 |
DQA1*006:01 |
DQB1*023:01 |
|
5 |
DRB1*015:02 DRB1*013:01 |
DQA1*BOX3 DQA1*BOX4 |
DQB1*058:01 DQB1*BOX4 |
|
6 |
DRB1*004:03 DRB1*BOX1 |
DQA1*014:01:1 DQA1*002:01 |
DQB1*BOX1 DQB1*BOX5 |
|
7 |
DRB1*015:02 DRB1*BOX2 |
DQA1*006:01 DQA1*001:01 |
DQB1*023:01 DQB1*002:01 |
|
8 |
DRB1*004:02 DRB1*BOX3 |
DQA1*014:01:1 DQA1*BOX4 |
DQB1*BOX1 DQB1*BOX2 |
|
9 |
DRB1*015:02 DRB1*BOX4 |
DQA1*006:01 DQA1*010:01 |
DQB1*015:01 DQB1*BOX3 |
|
10 |
DRB1*004:02 DRB1*BOX5 |
DQA1*014:01:1 DQA1*BOX4 |
DQB1*BOX1 DQB1*BOX5 |
|
11 |
DRB1*015:02 DRB1*BOX6 |
DQA1*006:01 DQA1*001:01 |
DQB1*058:01 DQB1*BOX4 |
|
12 |
DRB1*015:02 DRB1*004:01 |
DQA1*006:01 DQA1*002:01 |
DQB1*023:01 DQB1*015:01 |
Источник: составлено авторами.
Средний уровень полиморфизма характеризует ген DRB1 самоедской лайки (7 аллелей) и русского черного терьера (6 аллелей), а также генов DRB1, DQB1 и DQA1 боксера (9, 9 и 7 аллелей соответственно).
На основании анализа полиморфизма трех генов класса II локуса DLA можно заключить, что порода самоедская лайка и русский черный терьер оказываются генетически выровненными по генам DRB1 и DQA1, но полиморфными по гену DQB1. Что касается пород сибирский хаски и боксер, то гены DRB1, DQA1 и DQB1 у них относительно выравнены генетически, за исключением гена DRB1 у сибирского хаски. Можно предположить, что в случае аборигенных пород отечественного разведения сниженное разнообразие аллелей этих генов связано с изолированностью локальной популяции и сопутствующим инбридингом при разведении в ней собак. Особо следует выделить породу русский черный терьер, которая своим происхождением связана с российским питомником «Красная звезда».
В исследованиях, так же как и в работе зарубежных коллег, среди 85 из общего числа выявленных аллелей 40 аллелей были впервые охарактеризованы (табл. 1). Появление новых неохарактеризованных аллелей свидетельствует о высоком уровне полиморфизма генов DRB1, DQB1 и DQA1 локуса DLA. Из-за сложности депонирования новых аллелей в базе IPD-MHC дальнейшая характеристика и присвоение названий является задачей ближайшего будущего. Название новой аллели в данной работе включает название гена, сокращенное название породы собак и порядковый номер аллельного варианта в исследуемой выборке. В последующем планируется осуществить депонирование вновь выявленных аллелей в базе данных ISAG.
Для популяционных исследований информация о гаплотипах ГКГ обладает существенными преимуществами по сравнению с частотами аллелей одного локуса. Это связано с тем, что отсутствие рекомбинации между генетическими локусами в ГКГ обеспечивает поддержание определенных гаплотипов. В результате анализа гаплотипов был обнаружен 61 гаплотип в локусе DLA, при этом по 14 из них относятся к породе самоедская лайка и русский черный терьер, 16 гаплотипов принадлежат сибирского хаски, а 17 – боксеру (табл. 1). Анализ встречаемости разных гаплотипов в четырех исследуемых породах собак позволяет выделить гаплотипы, преимущественно встречающиеся у собак одной породы. Так, к породоспецифичным гаплотипам можно отнести DRB1*015:01-DQA1*006:01-DQB1*019:01, DRB1*040:01-DQA1*010:01-DQB1*019:01, DRB1*001:01-DQA1*003:01-DQB1*004:01, DRB1*015:02-DQA1*006:01-DQB1*023:01, которые характерны для собак породы самоедская лайка, сибирский хаски, русский черный терьер и боксер соответственно. Наименьшая частота встречаемости гаплотипов составила 0,05 для породы сибирский хаски, а для собак породы русский черный терьер, самоедская лайка и боксер частоты гаплотипов были одинаковые – 0,08. Существование определенных гаплотипов в отдельных породах может быть аргументировано благодаря хорошо известной информации о генетической структуре класса II локуса DLA, в котором 3 гена расположены в хромосоме 12 в непосредственной близости друг от друга, что значительно снижает частоту рекомбинаций между ними.
В настоящей работе не удалось выявить общих гаплотипов по аллелям трех генов класса II DLA среди собак всех четырех пород. Следует обратить внимание на высокую частоту встречаемости гаплотипа DRB1*001:01-DQA1*003:01 по двум генам в породе русский черный терьер – 9 из 14 гаплотипов для этой породы (табл. 1). Это может указывать на относительно молодой возраст породы, а также демонстрирует явление нарушения равновесия по сцеплению. Вновь выявленные аллели локуса DLA в отдельных породах собак в данном исследовании расширили круг гаплотипов по сравнению с уже известными. К ним можно отнести гаплотип DRB1*001:01-DQA1*003:01-DQB1*RBT4, который встретился у одной собаки породы русский черный терьер из 12, гаплотип DRB1*004:02-DQA1*014:01:1-DQB1*BOX1, встретившийся дважды в породе боксер из 12 исследованных особей.
Еще одной характеристикой генетической изменчивости четырех описываемых пород является показатель гетеро- и гомозиготности по генам DLA (табл. 2). Сравнение частоты гомозиготных собак разных пород по гену DRB1 демонстрирует, что максимальная частота отмечается в породе самоедская лайка (0,8), далее у русского черного терьера она составляет 0,5 и сравнима с этой частотой встречаемость гомозиготных собак в породе сибирский хаски (0,42). Наименьшая частота гомозиготных собак характерна для породы боксер (0,25). По гену DQA1 распределение гомозиготных собак по четырем породам полностью повторяет отмеченную картину для гена DRB1. Что касается гена DQB1, то распределение частот гомозиготных собак в разных породах резко отличается от предыдущих случаев. Не встретились гомозиготные собаки в породе самоедская лайка, сходная частота встречаемости гомозигот по этому гену была обнаружена для пород сибирский хаски и русский черный терьер (0,5–0,6). Показатель гомозиготности для собак породы боксер оказался неизменным (0,25). Объяснением наблюдаемой изменчивости по частотам гомозиготных собак в разных породах служат две причины: разная степень давления отбора внутри породы при разведении, а также время, прошедшее с момента создания породы.
С целью поиска связей между аллелями каждого из изученных генов, а также выяснения возможных межпородных связей аллелей генов DRB1, DQA1, DQB1 локуса DLA у собак пород самоедская лайка, сибирский хаски, русский черный терьер и боксер было проведено построение филогенетических деревьев, результаты которого приведены на рис. 3–5. Был использован метод Байеса, который позволяет определить вероятное дерево родственных связей между организмами на основе имеющихся данных относительно характеристик ДНК или белков, а также моделей эволюционных изменений. Построение филогенетических деревьев осуществлялось поэтапно, включая построение деревьев на основе аллельного разнообразия по каждому изучаемому гену в пределах отдельной породы. После этого были построены филогенетические деревья для каждого конкретного гена с привлечением данных по четырем изученным породам. Вероятные клады на филогенетических деревьях на рисунках отмечены овалами.
Филогенетическое дерево по нуклеотидной последовательности гена DRB1 для пород сибирский хаски, самоедская лайка, русский черный терьер и боксер представлено на рис. 3. Корнем этого дерева является аллель DRB1_00103. По результатам построения филогенетического дерева для аллелей гена DRB1 пород самоедская лайка, сибирский хаски, русский черный терьер и боксер, можно выделить две породы – сибирский хаски и боксер, хотя это не относится ко всем аллелям данного гена. Данное филогенетическое дерево характеризуется сложной картиной взаимоотношения аллелей гена DRB1, в то же время можно отметить породную специфику отдельных аллелей.
Филогенетическое дерево, построенное на основе нуклеотидной последовательности гена DQA1 для пород сибирский хаски, самоедская лайка, русский черный терьер и боксер, представлено на рис. 4. Корнем данного дерева является аллель DQA1*012012. Анализ данного филогенетического дерева не позволил продемонстрировать межпородное аллельное разнообразия по данному гену.
Филогенетическое дерево по нуклеотидной последовательности гена DQB1 для пород сибирский хаски, самоедская лайка, русский черный терьер и боксер представлено на рис. 5.
Корнем этого дерева является аллель DQB1_00803_H. Аллели DQB1_HAS2_H, DQB1_BOX1_B, DQB1_020_01_H, DQB1_029_01_H, DQB1_015_01_B и DQB1_RBT4_R занимают базальное положение на дереве. Четыре породы, проанализированные по аллелям данного гена, демонстрируют межпородные различия. Это позволяет выделить отдельные клады аллелей для каждой породы. В частности, в кладу для боксера попадают аллели DQB1_BOX2 и DQB1_BOX5, для самоедской лайки – DQB1_SAM4 и DQB1_SAM6, для сибирского хаски – DQB1_HAS3 и DQB1_HAS4, для русского черного терьера – DQB1_RBT3 и DQB1_RBT5.
Таблица 2
Частота встречаемости гомозигот и гетерозигот по генам DRB1, DQA1, DQB1
|
DRB1 |
DQA1 |
DQB1 |
|||
|
Частота абс. (отн.) |
Частота абс. (отн.) |
Частота абс. (отн.) |
|||
|
гомозиготы |
гетерозиготы |
гомозиготы |
гетерозиготы |
гомозиготы |
гетерозиготы |
|
Самоеды |
|||||
|
8 (0,80) |
2 (0,20) |
8 (0,80) |
2 (0,20) |
0 (0) |
10 (1) |
|
Хаски |
|||||
|
5 (0,42) |
7 (0,58) |
5 (0,42) |
7 (0,58) |
6 (0,50) |
6 (0,50) |
|
Русский черный терьер |
|||||
|
6 (0,50) |
6 (0,50) |
6 (0,50) |
6 (0,50) |
7 (0,50) |
5 (0,50) |
|
Боксер |
|||||
|
3 (0,25) |
9 (0,75) |
2 (0,17) |
10 (0,83) |
3 (0,25) |
9 (0,75) |
Источник: составлено авторами.
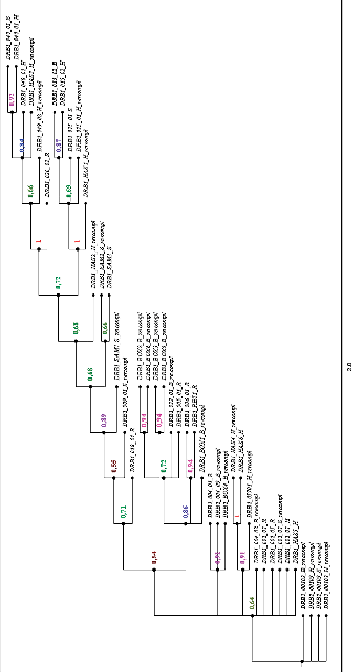
Рис. 3. Филогенетическое дерево гена DRB1 для представителей пород самоедская лайка, сибирский хаски, русский черный терьер и боксер Источник: составлено авторами по методу Байеса
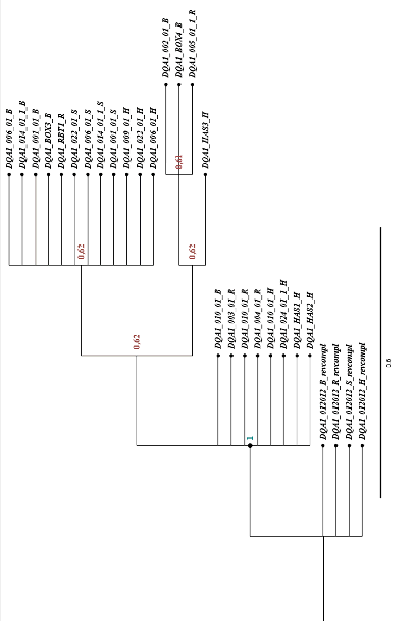
Рис. 4. Филогенетическое дерево гена DQA1 для представителей пород самоедская лайка, сибирский хаски, русский черный терьер и боксер Источник: составлено авторами по методу Байеса
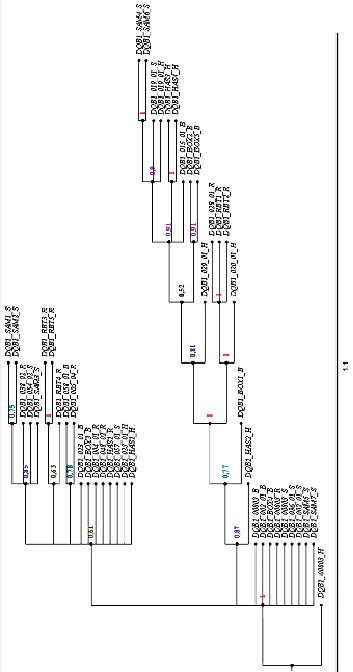
Рис. 5. Филогенетическое дерево гена DQB1 для представителей пород самоедская лайка, сибирский хаски, русский черный терьер и боксер Источник: составлено авторами по методу Байеса
Расширение объема выборки позволит в дальнейшем продолжить поиск филогенетических связей между породами на примере аллельного разнообразия генов локуса DLA.
Заключение
Проведенный анализ полиморфизма генов DRB1, DQA1 и DQB1 класса II DLA позволил сделать первый шаг в первичной характеристике существующего разнообразия пород собак на примере самоедской лайки, сибирского хаски, русского черного терьера и боксера российского разведения. В перспективе полученные данные могут способствовать прогнозированию риска развития у собак отдельных пород аутоиммунных заболеваний и канцерогенеза с учетом известных ассоциаций конкретных гаплотипов с определенными заболеваниями. Поиск филогенетических связей по аллелям генов локуса DLA расширит понимание общей картины эволюции вида Canis lupus familiaris – собаки домашней и происхождение отдельных ее пород.
Конфликт интересов
Благодарности
Финансирование
Библиографическая ссылка
Сафарова М.Х., Кан С.В., Душанова Г.А. АНАЛИЗ ГАПЛОТИПОВ ГЕНОВ ЛОКУСА DLA КЛАССА II У СОБАК РАЗНЫХ ПОРОД // Научное обозрение. Биологические науки. 2025. № 2. С. 81-91;URL: https://science-biology.ru/ru/article/view?id=1410 (дата обращения: 10.02.2026).
DOI: https://doi.org/10.17513/srbs.1410


 science-review.ru
science-review.ru